DNA 分子運算法
DNA 計算法介紹 (Introduction of DNA computing)
摘要:電腦已與人類的生活息息相關,密不可分。電腦的功能也是日新月異,一躍千里。然而,當碰上某些組合性問題時,即令是超級電腦,有著極快速的運算能力,也得耗時經年,方能解出答案。在尋找替代運算方法的努力過程中,DNA 序列的多樣性和專一性,以及其相關之分子生物技術,已被證明能夠用於運算該類組合性問題,並具有優於電腦的潛在能力。本文即對DNA 運算法的由來、其優點和缺點、以及未來的展望,作一概括的介紹。
背景:電腦在處理一個運算問題時,其效率是由軟體及硬體所共同決定的。比如說,有一等加級數如下:
S(n) = 1 + 2 + 3 + ..... + n
在此,n 除了是一個正整數之外,其值亦表示該級數所含之項數,也就是此問題的大小 (problem size)。若按照題意,用最直接了當的方式來解此題目,電腦得進行 (n-1) 個加法運算。此時,求得解答所需之運算量及所耗費的時間,會與問題的大小 n 成正比。然而,若能利用等差級數特有的性質來解題,即其和等於 (前項 + 後項) × 項數 ÷ 2,則不論問題的大小 n 有多大,求得解答所需之運算將永遠是一個加法、一個成法、及一個除法。除了使用不同的算式之外,平行處理的硬體架構也可以用來增進電腦解題的效。如同上之問題,當有兩個處理器 (cpu) 可供使用時,每個處理器可負責一半的運算量。此時總運算量並無改變,但獲得解答所須的時間,理論上將縮短一半。
然而,在真實世界裡,問題的運算之於電腦卻非盡如上述那般容易。首先,不是所有問題都有簡潔的運算法。比如有一問題,其運算量與問題大小 n 之間,只存在著 2n 的指數關係。此時,若 n = 10,則解得答案所須的運算量為 1024。但若問題的大小放大十倍成 n = 100,運算量則要膨脹到 1.27 × 1030。此時縱有一台每秒能處裡 1× 1010 個運算的電腦,也得耗費 4× 1012 年,方可完成這種天文數字般的工作量。雖然,同時使用多個處理器,能夠縮短解題的時間,惟以現有之科技及電腦之硬體架構,無法造出一台足以在有限時間內,解答上述例題之平行電腦。
關於這類「困難」的問題,因其總運算量無法有效縮減,硬體架搆的改良甚至從新設計,遂成為科學家們努力的方向。而 DNA 計算器的發明算,就是在這樣的條件和背景之下,所得到的突破性進展。第一部實際建構並執行解題運算的 DNA 計算器,是由美國的 Leonard Adleman 博士在 1994 年所完成 (Adleman 1994, Molecular computation of solutions to combinational problems. Science 266, 1021-1024)。Adleman 博士利用 DNA 序列的多樣性及其互補時的專一性,將資訊儲存於 DNA 片段,再用時下之分子生物技術來「處理」該資訊。藉由多量但細微的分子,在極小的體積裡進行快速的生化反應,造就成目前電腦所望塵莫及的巨大平行處理能力。Adleman 博士用他所設計出來的 DNA 計算器解出了一道叫作「漢米爾頓途徑 (Hamiltonian Path)」的運算問題。漢米爾頓途徑為「非確定性多項式問題 (NP-complete problem)」的一種。本質上,找出該問題的答案所需之運算量,與問題大小 n 為非線性關係。與漢米爾頓途徑相似的另一個「非確定性多項式問題」為漢米爾頓迴路 (Hamiltonian Cycle) 。以下我們即以漢米爾頓迴路為例,來說明如何用DNA計算問題。
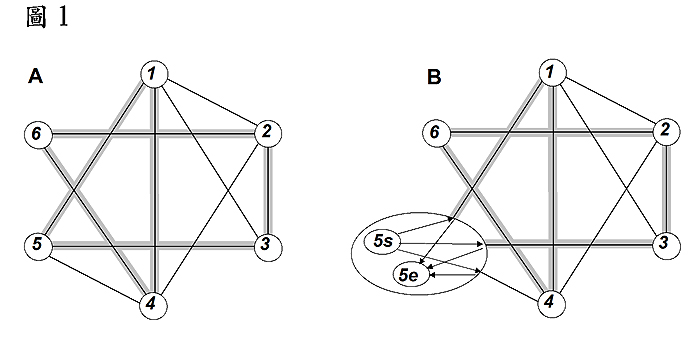
圖1: (A) 一個由6個點及若干代表雙向路徑之線條所構成的圖形。該圖形中有一漢米爾頓迴路,即灰色線條之部份。 (B) 在執行DNA 運算之第一步驟時,點5被選來當作起迄點。在往後的運算中,該點將被視為兩個獨立的點,一為起點5s,另一個為迄點 5e。
例題:如圖1A所示,有一圖形包含六個點,以不同之阿拉伯數字標示。此外,若干點與點之間有線條相接連。每條線代表一雙向途徑,為點與點之間的連絡道路。而所謂的漢米爾頓迴路,即指此類圖型中,經過所有的點,且每一點只經過一次之簡單迴路。解答此一問題時,首先要決定圖型中是否存有漢米爾頓迴路。如果有,則進一步指出該迴路。以分子生物技術解題時,其運算流程可歸納如下:
1. 隨機選取一點,如圖1A之點5,並以此點為起點及迄點。
2. 完成圖形中,所有可能之路徑組合。
3. 起迄點為點5之路徑組合留下待用,其餘之路徑組合捨棄。
4. 存留之路徑組合中,路過之點數與圖形所含有之點數相同者留下,其餘捨棄。
5. 存留之路徑組合中,每個點都經過者留下,其餘捨棄。
6. 至此,若沒有路徑組合存留,則此圖型不含漢米爾頓迴路。運算到此為止。
7. 若有路徑組合存留,則此圖型含有漢米爾頓迴路。指出該迴路。
現在我們來看詳細的實驗內容及其所代表之運算含意。首先,在運算流程之第一步驟,點5被選作解題過程之起迄點。在往後的運算中,該點將被視為兩個獨立的點,一為起點5s (表5 start),另一個為迄點 5e (表5 end) (圖1B)。
在執行運算流程之第二步驟前,要先把問題圖形之資訊編碼成DNA序列。首先,用含有20個鹼基的DNA片段 (oligonucleotide) 來代表圖形中的點;每一個點有它對應的一條DNA片段,且每一條DNA片段的鹼基序列皆不相同。如圖2A所示,O2 代表點2 (此處大寫英文字母O 表Oligonucleotide,阿拉伯數字指所代表之點),O3代表點3,O6代表點6。因DNA 可為雙股結構,其互補之一股用符號 Ø 來表示,如與O2互補之DNA片段為Ø2。接著,分別用兩條DNA片段來代表圖形中之每一條線。例如,在圖1中,點2和點3之間有一線條相連,O2-3及O3-2則用來代表由點2行至點3及由點3行至點2之路徑。O2-3及O3-2同樣是由20個鹼基所組成。然而,O2-3之5’端的10個鹼基序列等同於O2之3’端的10個鹼基序列,其3’ 端的10個鹼基序列則等同於O3之5’端的10個鹼基序列。同理,O3-2為O3之3’端的10個鹼基序列及O2之5’端的10個鹼基序列所組成 (圖2A)。圖形裡其餘之線條及其對應之DNA片段皆以此模式組成。又,因與5s及5e相連的線已成為單向路徑,故只用一條DNA片段來表示。

圖2:圖形裡的點和線都用oligonucleotide 予以編碼。(A) 由20個鹼基所組成的DNA 片段O2代表點2,與其互補之另一股DNA片段則為Ø2。代表由點6走到點2之路徑則由O6 之3’端的10個鹼基序列及O2之5’端的10 個鹼基序列組合而成。圖形裡其餘的點和線,也按此規則,由20個鹼基,組成序列互不相同之DNA片段來代表。(B) 單股DNA 片段可依其互補之特性,對應接續,並在DNA ligase的黏合作用後,形成更長的分子。箭頭指處即DNA ligase 作用之處。
此時,整個圖形的基本資訊已編寫並儲存在一組DNA片段裡。接著,把所有的Øx 及Ox-y (x 及 y 表不同之點 ) 置於同一試管。藉由序列互補的性質,Øx會把可能的Ox-y組合起來。如圖2B所示,Ø2可以橋接O6-2及 O2-3。同理,Ø6 可以橋接O4-6 及 O6-2。如此一來,一段代表由點4 走到點3之路徑組合,即O4-6 : O6-2 : O2-3 ,便可形成。 之後,再以DNA ligase ,將組合好的Ox-y,黏接成單一之長序列DNA分子。不同的Ox-y,經過排列組合,所形成之長條DNA分子,會有不同之鹼基序列。而其所代表的,即為此圖形中,所有可能之路徑組合。
運算流程之第三步驟,是用上一步驟所得之長序列DNA分子為模板 (template),O5s及Ø5e為引子 (primers) ,來執行聚合脢鏈鎖反應 (PCR)。被放大的DNA分子,其雙股螺旋之一股必以O5s為其5’端,並以O5e為其3’端。也就是說,被放大的DNA分子,其所代表的路徑,皆以5s 為起點,並以5e為迄點。
按題意,漢米爾頓迴路須經過圖形裡的每一個點,而解題運算的第一步驟已將點5拆成起、迄兩個不同的點,故例題圖形之漢米爾頓迴路須包含七個點。又,每一個點是由帶有20個鹼基的DNA分子所代表,故經過七個點的路徑組合,其DNA分子應有140個鹼基對。是以在運算流程之第四步驟,其執行的方式可利用膠體電泳 (agarose gel electrophoresis), 將上一步驟PCR所得的產物,依DNA分子的大小 (長短) 分離。長度為140bp之DNA 留下,其餘丟棄。
運算流程之第五步驟,旨在驗證長度為140bp之DNA分子,的確經過圖形裡所有的點,而非重覆某點而遺漏另一點。執行的方式可以是親和性純化法(affinity purification)。例如,將上一步驟所得之140bp DNA 分子加熱分離成單股 (denature),接著通過帶有Ø1之層析管住 (column)。如此,含有O1的單股DNA會吸附於管住,不含O1者則流失丟棄。附著之140-bases DNA 可以用鹼性容液洗出,中和後再通過帶有Ø2之層析管住。如此循還重覆直至完成對所有點的檢驗。由於前面已確認過,140bp DNA 所代表之路徑起迄點分別為5s 及 5e,故對O5s及O5e的檢測可略去不作。
至此,若無DNA 存留,表示題目之圖形中,不存在有漢米爾頓迴路,運算到此結束。如有DNA 存留,則該存留之DNA 內藏漢米爾頓迴路,運算繼續。圖1A 的例題中實含有一漢米爾頓迴路,所以要進行最後一步的運算。
最後一步的運算,在揭露隱含在DNA 序列裡的漢米爾頓迴路,也就是該迴路的行進途徑,或者說該迴路經過各點的順序。已知起點為5s,終點為5e,故順序應為5s à v à w à x à y à z à 5e。要求得答案,可分別用O5s及Ø1、O5s及Ø2、O5s及Ø3、O5s及Ø4、O5s及Ø6 五組引子對(primer pairs) ,同時進行PCR。以圖1A 為例,O5s及Ø1 會放大出兩條DNA 片段,其長度分別為 40bp 及 120bp。這個結果表示點1 可以和點5s相鄰,或被五個點所隔開。亦即點1可以是在 v ,也可以是在 z 的位置。同理,O5s及Ø2 也會放大出兩條DNA 片段,其長度分別為 60bp 及 100bp,代表點2可以是 w ,也可以是在 y 的位置。要知道,答案本為一雙向之迴路。因解題之需要,將其改為單向之路徑,是以出現兩個可能的位置。在完成此輪 PCR 後,可以得到如下結果:
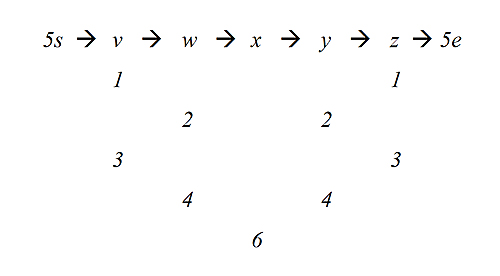
此時尚無法辨認5s à1 à之後是接點2還是點4。只須分別再以O1及Ø2 、O1及Ø4作一輪PCR ,點1與點 2及點4在路徑上之相關位置即可得知。在圖1A 之例題中,以O1及Ø4 為引子所作之PCR,其產物為一40bp 之DNA 片段,故知點1與點4在漢米爾頓迴路上相鄰。至此,此例題之答案解出如下:
討論:利用DNA與分子生物技術來執行資訊運算的工作,具有低耗能及高資料儲存密度的優點。然而,DNA運算法最有價值的地方,是它的巨大平行處理能力,以及資料的篩選方式。就前面所舉的漢米爾頓迴路問題為例,在迷宮似的圖形裡,電腦必須使用嘗試錯誤的策略,對每一條可能的路徑組合,循序走過,始能求得答案。對一個只有六個點的圖形來說,電腦可以在彈指之間,完成解題所須的運算。但是,當圖形的點數放大100倍或1000倍後,電腦可能得花好幾年的時間,才能走完所有可能的路徑組合,並決定出圖形中是否有題目所要的答案。相形之下,應用DNA計算法,無論圖形之中有多少點,所有可能之路徑組合都將在一個步驟內完成。隨後,在巨量的資料庫裡,直接了當的將答案撿選出來。
當然,DNA 運算法並非沒有缺點。最顯而易見的部份是,在實驗室裡進行生化反應是耗時費事的。因此,即令DNA計算法可以提供巨大之平行處理能力,除了當問題的大小n大於一定之值時,用DNA計算法是不經濟的。其次,在真正進行巨量運算時,DNA計算法所可能面對的實驗障礙,譬如DNA分子的穩定性和序列忠實性對結果所可能造成之誤差,至今上尚未被實際驗證過。還有,目前已被提出的DNA運算法,皆不具備普遍通用之性質,只能用於極少數之題型。
顯然的,DNA 運算法尚未達到一成熟的階段,許多的研發工作仍有待進行。在此同時,我們實也不應將眼光侷限在DNA分子。生命現象及生物分子的多樣多變性,都有可能被用來作為處理資訊及解答問題的工具。有朝一日,利用一個細胞之整體生理生化反應的運算法,或可被發展出來。此外,在研究生物運算法的過程中,除了可以解決資訊科學的若干問題,實際上也能幫助我們對複雜的生命體有更進一步的認識。在這個屬於生物技術的世紀,在許許多多物種的基因體被定序等待解碼的同時,運用生物既存的方式與能力來解開是項奧密,亦是個值得投入思考與研究的方向。
作者 吳少傑
以上原文刊載於 生物科技雜誌 第九期 2003 年 二月號 59-36頁